¡Atención!
Estudio Computacional
La comunidad de investigación se ha movilizado rápidamente para combatir la actual pandemia del SARS-CoV-2, basándose en años de trabajo sobre el virus del SARS-CoV anterior. La proteína de espiga de este virus será una figura central en esta lucha, ya que es el objetivo principal de los anticuerpos que proporcionan inmunidad contra el virus. Las superficies de los coronavirus están cubiertas con estas espigas, dándoles su aspecto distintivo de corona en micrografías electrónicas. Las espigas inician el proceso de infección, se unen a los receptores y luego se fusionan con la membrana celular para liberar el genoma viral en su interior. Muchos otros virus envueltos usan proteínas similares a espigas para infectar las células, incluida la hemaglutinina de la gripe y las glucoproteínas de la envoltura del VIH-1 y el ébola.
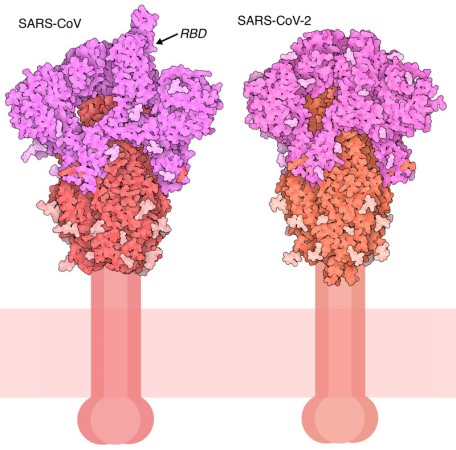
Proteína de pico de SARS-CoV, con un dominio de unión al receptor (RBD) en la posición superior, y una conformación cerrada del pico de SARS-CoV-2. El fragmento S1 se muestra en magenta y el fragmento S2 en rojo, con glucosilación en tonos más claros.
La proteína espiga se compone de tres cadenas idénticas, que juntas forman un complejo con un pequeño dominio dentro del virus, un segmento que abarca la membrana y un gran ectodominio (porción de un polipéptido en una membrana celular que va más allá de la membrana hacia el espacio extracelular) que se extiende hacia afuera del virus. Además, la espiga es una glicoproteína: el ectodominio está cubierto con cadenas de azúcar que ayudan a enmascarar el virus del sistema inmunitario. Las estructuras de los picos SARS-CoV y SARS-COV-2 que se muestran aquí (entradas PDB 6crz y 6vxx) incluyen solo el ectodominio y, como puede se puede ver, son muy similares. Cada cadena se sintetiza en una sola pieza, pero luego se corta mediante proteasas celulares en dos piezas funcionales. El fragmento S1 externo, de color magenta, se une a los receptores celulares, y el fragmento S2, de color rojo, dirige la fusión del virus con la célula. Ambas estructuras incluyen solo porciones de las muchas cadenas de azúcar que recubren la espiga, ya que los azúcares son muy flexibles y difíciles de observar.
Ver archivo PDB en el simulador 3D
Ver archivo PDB en el simulador 3D
Las estructuras recientes han revelado que las proteínas espiga de SARS-CoV y SARS-CoV-2 son bastante flexibles. En estas estructuras, los dominios de unión al receptor se ven en diferentes conformaciones. A menudo, solo un dominio se extiende hacia arriba, pero a veces todos están escondidos o extendidos. La conformación extendida es necesaria para unirse a los receptores, por lo que esta flexibilidad es una gran ventaja para el virus. Los investigadores plantean la hipótesis de que las variantes recientes del SARS son virulentas porque sus dominios de unión al receptor son particularmente flexibles, mientras que los coronavirus más extendidos que causan el resfriado común son menos peligrosos porque son menos flexibles.
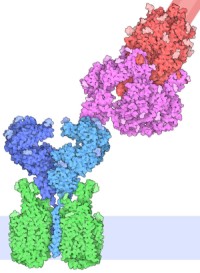
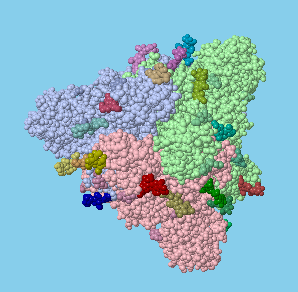
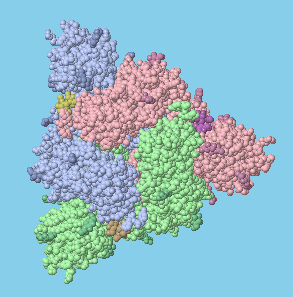
Ilustración de un complejo de la proteína espiga (roja y magenta) unida a su receptor ACE2 (azul). ACE2 es parte de un complejo con el transportador de aminoácidos B0AT1 (verde). La membrana celular se muestra esquemáticamente en azul claro en la parte inferior.
La proteína de espiga se une a ACE2 (enzima convertidora de angiotensina 2) en la superficie de las células. ACE2 es una enzima que activa la angiotensina, una hormona peptídica involucrada en el control de la presión arterial. La ACE2 se encuentra en las células pulmonares, cardíacas, renales e intestinales, lo que hace que estas células sean el objetivo de la infección por el virus. La entrada PDB 6m17 revela el complejo de ACE2 con el dominio de unión al receptor de la espiga de SARS-CoV-2. En el complejo, ACE2 también está unido al transportador de aminoácidos B0AT1. La ilustración se creó superponiendo este complejo con la estructura de la proteína espiga en la entrada PDB 6vsb.
Ver archivo PDB en el simulador 3D
Ver archivo PDB en el simulador 3D
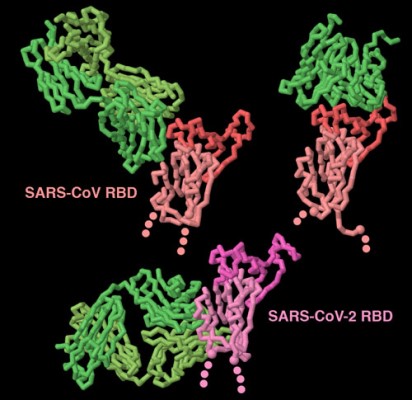
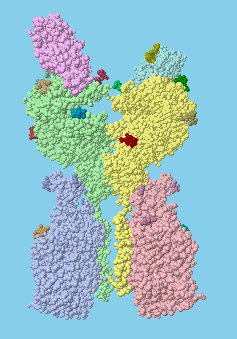
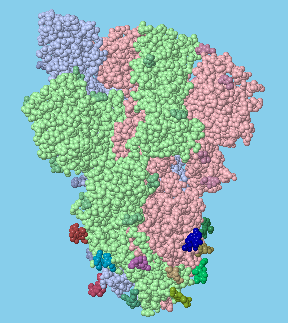
Nuestro sistema inmunitario se defiende cuando los coronavirus nos infectan. La espiga es el objetivo principal de esta protección, ya que está expuesta en la superficie del virus. Estas tres estructuras (entradas de PDB 3bgf, 2ghw y 6w41 muestran que los anticuerpos (mostrados en verde en la imagen superior) pueden reconocer las proteínas de espiga de muchas maneras. Dos de estos anticuerpos bloquean la porción de unión al receptor del dominio (mostrado aquí en colores más brillantes), pero el otro anticuerpo se dirige a un sitio escondido en la base del dominio, que solo se expone cuando el anticuerpo se une a él. Para explorar estas estructuras con más detalle, haga clic en la imagen para obtener un JSmol interactivo.
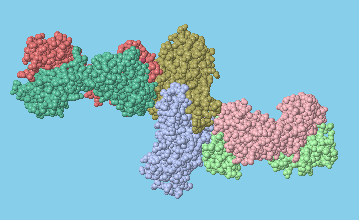
Ver archivo PDB en el simulador 3D. Estructura cristalina de rayos X del dominio de unión al receptor de espiga de coronavirus SARS en complejo con Fab F26G19 (anticuerpo recombinante de ratón).
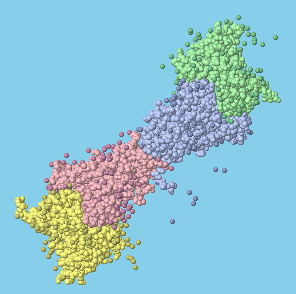
Ver archivo PDB en el simulador 3D
Estructura cristalina del dominio de unión al receptor de la proteína del pico de SARS en complejo con un anticuerpo neutralizante, 80R
Un anticuerpo neutralizante (NAb) es un anticuerpo que defiende una célula de un patógeno o partícula infecciosa neutralizando cualquier efecto que tenga biológicamente. La neutralización hace que la partícula ya no sea infecciosa o patógena.Los anticuerpos neutralizantes son parte de la respuesta humoral del sistema inmune adaptativo contra virus, bacterias intracelulares y toxina microbiana. Al unirse específicamente a las estructuras superficiales (antígeno) en una partícula infecciosa, los anticuerpos neutralizantes evitan que la partícula interactúe con sus células huésped que podría infectar y destruir. La inmunidad debida a anticuerpos neutralizantes también se conoce como inmunidad esterilizante, ya que el sistema inmunitario eliminó la partícula infecciosa antes de que ocurriera cualquier infección.
Ver archivo PDB en el simulador 3D. Estructura cristalina del dominio de unión al receptor SARS-CoV-2 en complejo con el anticuerpo humano CR3022
El anticuerpo CR3022 original se generó secuenciando linfocitos de sangre periférica de un paciente expuesto al SARS-CoV. Se demostró que este anticuerpo neutraliza el SARS-COV en una acción concertada con el clon CR3014. La presencia de ambos anticuerpos proporciona una acción de bloqueo de la interacción SARS-COV RBD-ACE2.
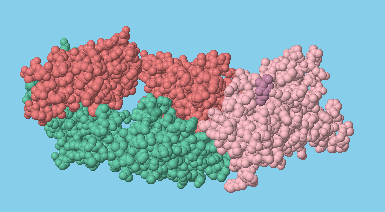
Anticuerpos unidos al receptor de la espiga
Ver archivo PDB en el simulador 3D.
Ver archivo PDB en el simulador 3D
Ver archivo PDB en el simulador 3D
Dominio de unión del receptor de la espiga (magenta o rosa) con anticuerpos (verde). Usa los botones para comparar las tres estructuras.