Motivos y Dominios
Lic. Raúl Hernández M.
Contenidos:
1. Introducción2. Plegamiento
2. Plegamiento Anómalo
3. Motivos
β-α-β
Barril β
α-giro-α
Mano EF
β-giro-β
Plegamiento de Rossmann
Cremallera
Llave Griega
4. Desnaturalización
Desnaturalización de 1enh
Proteína Desnaturalizada (1ztr)
Comparación entre 1ehn y 1ztr
5. Dominios
Dominios en troponina C
Dominios en piruvato quinasa
Introducción
De lo que ahora sabemos sobre las estructuras terciarias de miles de proteínas globulares, está claro que la mioglobina ilustra solo una de muchas maneras en el que una cadena polipeptídica puede plegarse.
En la siguiente tabla se muestra las proporciones de conformaciones α y hélice β (expresadas como porcentaje de residuos en cada tipo) en varias proteínas pequeñas globulares y monocatenarias. Cada una de estas proteínas tiene una estructura distinta, adaptada para su función biológica particular, pero juntas comparten varias propiedades importantes con la mioglobina. Cada uno está plegada compactamente y en cada caso las cadenas laterales de aminoácidos hidrofóbicos son orientadas hacia el interior (lejos del agua) y el lado hidrofílico las cadenas están en la superficie. Las estructuras también están estabilizadas por una multitud de enlaces de hidrógeno y algunas interacciones iónicas.
| Residuos Totales | Hélice α | Hoja β |
|---|---|---|
| Quimotripsina (247) | 14 | 45 |
| Ribonucleasa (124) | 26 | 35 |
| Carboxipeptidasa (307) | 38 | 17 |
| Citocromo C (104) | 39 | 0 |
| Lisozima (129) | 40 | 12 |
| Mioglobina (153) | 78 | 0 |
Plegamiento
Principios básicos de la química que impulsan el plegamiento de proteínas
Las proteínas son moléculas grandes que se sintetizan en el entorno polar y acuoso de la célula. Se hacen uniendo aminoácidos en una secuencia particular. Debido a que cada uno de los 20 aminoácidos tiene una forma y unas propiedades químicas diferentes, las proteínas se pliegan en diferentes formas tridimensionales siguiendo los principios básicos de la química.
Embudo PlegableLas proteínas no parecen plegarse de una manera ordenada y gradual. Esto tomaría demasiado tiempo, dada la larga longitud de las cadenas de proteínas típicas y las muchas conformaciones que potencialmente pueden adoptar. En cambio, todo sucede a la vez, y una proteína puede alcanzar su forma final a través de muchas vías diferentes. Este proceso ha sido denominado un "embudo plegable", con muchas conformaciones intermedias diferentes, todas convergiendo hacia una estructura final. El truco del diseño de proteínas es descubrir las reglas que conducen a este tipo de embudo energético, sin tener conformaciones que se atasquen en pliegues inadecuados.
En la estructura final después de haberse llevado a cabo el proceso de plegamiento, los aminoácidos hidrofóbicos se ubican en la parte interior de la proteína y los aminoácidos polares con con carga, se ubican hacia la parte exterior, en donde están en contacto con el agua.


Plegamiento Anómalo
El plegamiento de una proteína es un proceso complejo que a veces puede provocar moléculas inadecuadamente plegadas. Estas proteínas mal plegadas suelen etiquetarse y degradarse dentro de la célula. Sin embargo, este sistema de control de calidad no es perfecto y pueden acumularse agregados intracelulares o extracelulares de proteínas mal plegadas, en particular a medida que la persona envejece. Depósitos de proteínas mal plegadas se asocian con una serie de enfermedades, entre ellas la amiloidosis.
Amiloidosis
El plegamiento anómalo de las proteínas puede producirse de manera espontánea o estar causado por una mutación en un gen concreto, que produce luego una proteína alterada. Además, algunas proteínas aparentemente normales, después de una escisión proteolítica anómala, pueden adoptar un estado conformacional único que induce la formación de conjuntos proteicos fibrilares largos consistentes en láminas β plegadas. La acumulación de estas proteínas insolubles que se agregan de manera espontánea, denominadas amiloides, se ha implicado en enfermedades degenerativas, como el Parkinson y la enfermedad de Huntington, y en particular en el trastorno degenerativo que constituye la enfermedad de Alzheimer.
El componente dominante de la placa de amiloide que se acumula en la enfermedad de Alzheimer es el amiloide β (Aβ), un péptido extracelular que contiene más de 40 a 42 residuos de aminoácidos. La cristalografía de rayos X y el espectroscopio de infrarrojos demuestran una conformación en lámina β plegada (color amarillo) característica en fibrillas no ramificadas.
Cuando estas fibrillas se forman en el cerebro, contribuyen al desarrollo de la enfermedad de Alzheimer. La fibrilla amiloide que se muestra aquí, la entrada PDB 2m4j, se aisló de un paciente con enfermedad de Alzheimer y muestra cuántos péptidos pequeños se acumulan para formar una fibrilla triple simétrica.
Fibrilla β amiloide de 40 residuos (PDB 2m4j. Puentes de hidrógeno
Péptido amiloide de 40 residuos Puentes de hidrógeno
Motivos
Para comprender una estructura tridimensional completa, debemos analizar sus patrones de plegado. Comenzamos definiendo dos términos importantes que describen patrones estructurales de proteínas o elementos en una cadena de polipéptidos y luego recurrir a las reglas de plegamiento
El primer término es motivo, también llamado pliegue o (más raramente) estructura supersecundaria. Un motivo o pliegue es un patrón de doblado reconocible involucrando dos o más elementos de estructura secundaria y la(s) conexión(es) entre ellos. Un motivo puede ser muy simple, como dos elementos de estructura secundaria plegada una contra la otra, y representan solo una pequeña parte de una proteína Un ejemplo es un giro β-α-β. Un motivo también puede ser un estructura muy elaborada que implica puntajes de segmentos de proteínas plegados, como el barril β.
β-α-β
Esqueleto
Barril β

Esqueleto
Otros Motivos
α-giro-α

Esqueleto
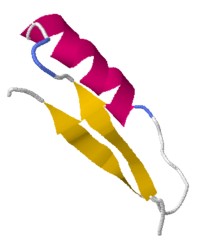
Mano EF
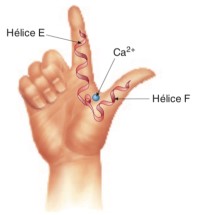
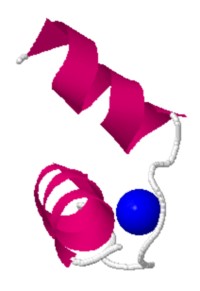
Esqueleto Interacciones de Ca+2 con cadenas laterales
β-giro-β, horquilla (gancho de pelo) β


Esqueleto
Plegamiento de Rossmann
Es un caso especial de la unidad ß-α-ß, pues consiste en dos o más subunidades ß-α-ß adyacentes. Es frecuente en proteínas que se unen a los nucleótidos
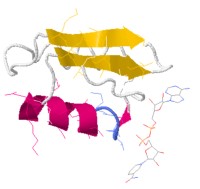
Esqueleto
Cremallera
consiste en los dos segmentos peptídicos en la hélice α, que a su vez se enrollan levemente uno alrededor del otro (formando así una espiral enrollada). En un extremo, estas hélices interactúan entre sí, y en el otro extremo interactúan con secuencias específicas de bases en el mayor surco del ADN.

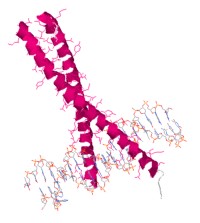
Esqueleto
La interacción entre las dos hélices se mantiene principalmente gracias a la interacción entre las cadenas laterales de leucina, localizadas cada 7 residuos en la secuencia (es decir, casi 2 vueltas de la hélice alfa).
Llave Griega
Cuando una lámina antiparalela se dobla sobre sí misma en un patrón que se asemeja a un diseño de alfarería griega, el motivo estructural se denomina llave griega.

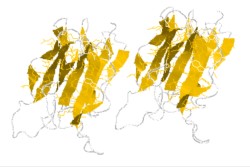
Esqueleto
Desnaturalización
Las proteínas llevan una existencia sorprendentemente precaria. Como hemos visto, la conformación protéica nativa es solo marginalmente estable. Además, la mayoría de las proteínas deben mantener flexibilidad conformacional para funcionar. El continuo mantenimiento del conjunto activo de proteínas celulares requerido en un conjunto dado de las condiciones se llaman proteostasis. Proteostasis es el término que se ha ocupado para definir a la homeostasis proteínica dentro de la célula. Este concepto abarca el estudio de las proteínas de manera individual, desde su síntesis, ubicación celular y función, hasta su vida media y degradación. Sin embargo, la proteostasis también se enfoca en la actividad grupal de las proteínas, analizando cuales de ellas funcionan dentro de una red de trabajo. Cuando las proteínas interactúan como una red de trabajo, producen flujos de información a través de diversos componentes. La eficiente coordinación entre las proteínas que conforman el flujo de la información es esencial para el correcto funcionamiento y supervivencia de la célula.
la vida de una proteína abarca mucho más que su síntesis y posterior degradación. La estabilidad marginal de la mayoría las proteínas pueden producir un equilibrio tenue entre los estados plegados y desplegados. Como las proteínas se sintetizan en los ribosomas, deben doblarse en sus conformaciones nativas. A veces esto ocurre espontáneamente, pero más a menudo requiere la asistencia de enzimas especializadas y complejos llamadas chaperonas. Muchos de estos mismos auxiliares de plegado funcionan para replegar proteínas que se vuelven transitoriamente desplegadas. Proteínas que no están correctamente dobladas a menudo tienen superficies hidrofóbicas expuestas que las vuelven "pegajosas", lo que produce la formación de agregados inactivos. Estos agregados pueden carecer de su función normal pero no son inertes; su acumulación en las células produce enfermedades que van desde la diabetes a la enfermedad de Parkinson y la enfermedad de Alzheimer. No es sorprendente que todas las células tengan vías elaboradas para el reciclaje y / o proteínas degradantes que están mal plegadas irreversiblemente.
Desnaturalización de la proteína 1enh
Modelo de la de la proteína 1enh de la Drosophila melanogaster o mosca de la fruta. Es una proteína de 3 hélices alfa de 61 residuos (H1, H2, H3).
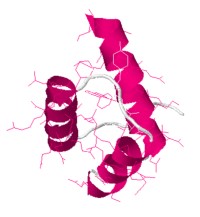
Puentes de hidrógeno
Esqueleto
Aminoácidos Hidrofóbicos, se ubican en el interior de la proteína
Bloques
Proteína desnaturalizada por una mutación (1ztr)

Una mutación de leucina a alanina cambia la cadena lateral de un hidrófobo más grande a uno más pequeño, lo que disminuye las interacciones de van der Waals y disminuye la estabilidad de la proteína.
Los cambios en la fuerza iónica hacen que la proteína mutante (1ztr) se doble y se despliegue reversiblemente. A elevadas resistencias iónicas, la proteína mutante existe en su estado nativo plegado, pero a la fuerza iónica fisiológica (~ 150 mM) la proteína se desnaturaliza.
Proteína Desnaturalizada por una mutación (pdb, 1ztr)
Puentes de hidrógeno. Mientras que el núcleo se interrumpe en el estado desnaturalizado, la estructura helicoidal y el viraje que conectan H2 y H3 permanecen intactos.
Esqueleto, el estado desnaturalizado es más grande, más elongado, y menos compacto que el estado nativo.
Bloques. H1 es más larga, por lo menos un giro comparada con el estado nativo.
Aminoácidos Hidrofóbicos, se ubican en el interior de la proteína
Proteína nativa y desnaturalizada sobrepuestas
Proteína nativa (1enh) mostrada en color azul y proteína desnaturalizada (1ztr) mostrada en color púrpura.
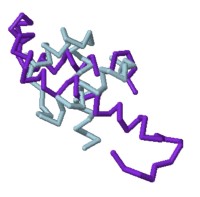
proteína nativa proteína nativa
proteína desnaturalizada proteína desnaturalizada
Bloques
Puentes de hidrógeno
Dominios
El segundo término para describir patrones estructurales es dominio. Un dominio, como lo definió Jane Richardson en 1981, es parte de una cadena polipeptídica que es independientemente estable o podría sufrir movimientos como una sola entidad con respecto a la proteína completa. Polipéptidos con más de unos cientos de residuos de aminoácidos a menudo se pliegan en dos o más dominios, a veces con diferentes funciones. En muchos casos, un dominio de una proteína grande retendrá su estructura tridimensional nativa, incluso cuando está separada (por ejemplo, por escisión proteolítica) del resto de la cadena polipeptídica. En un proteína con múltiples dominios, cada dominio puede aparecer como lóbulo globular distinto. Diferentes dominios a menudo tienen distintas funciones, como la unión de pequeñas moléculas o la interacción con otras proteínas. Las proteínas pequeñas usualmente tienen un dominio (el dominio es la proteína).
Las proteínas varían en peso molecular de mil a más de un millón de daltons. Es tentador pensar que el tamaño de estructuras globulares únicas plegadas aumentaría con el peso molecular, pero esto no es lo que se ha observado. Hay proteínas compuestas de aproximadamente 250 aminoácidos o menos y a menudo tienen una forma globular simple y compacta. Sin embargo, proteínas globulares más grandes se componen generalmente de dos o más estructuras reconocibles y distintas, denominadas dominios o módulos: estructuras de proteínas plegadas y compactas que generalmente son estables por sí mismas en solución acuosa.
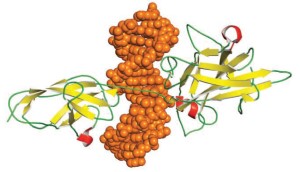
Un dominio es un término más genérico que designa una región de una proteína con interés biológico funcional o estructural. También se llama dominio a una región de la estructura tridimensional de una proteína con una función concreta, que incluye regiones no necesariamente contiguas en la secuencia de aminoácidos.
Ton-EBP es una proteína de unión a ADN que consiste en de dos dominios distintos. El dominio N-terminal se muestra aquí a la derecha, con ADN (naranja) en el medio, y el dominio C-terminal a la izquierda (pdb 1imh).
Dos dominios
Otros ejemplos de dominios
Troponina C
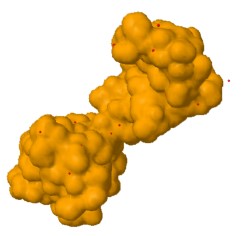
Dos dominios en la troponina C (pdb 4tnc)
Piruvato Quinasa
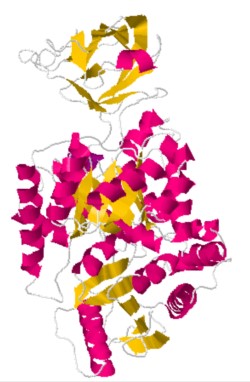
Tres dominios en la piruvato quinasa (pdb 1pkm)